该方法可以从合成聚合物数据存储中更有效地检索数据
越来越多的数据需要存储,而且通常需要长时间存储。合成聚合物是传统存储介质的替代品,因为它们可以保存存储的信息,同时占用更少的空间和能源。然而,通过质谱法检索数据会限制单个聚合物链的长度,从而限制其存储容量。
在《应用化学国际版》杂志上,研究人员介绍了一种克服这一限制的方法,允许直接访问特定位而无需读取整个链。
数据不断积累,包括业务交易、流程监控、质量保证或产品批次跟踪。几十年来存档这些数据需要大量空间和能源。对于需要不频繁访问的大量数据的长期存档,具有确定序列的大分子(如 DNA 和合成聚合物)是一种有吸引力的替代方案。
合成聚合物比 DNA 具有以下优势:合成简单、存储密度更高、在恶劣条件下稳定。它们的缺点是聚合物中编码的信息需要通过质谱(MS) 或串联质谱测序 (MS2) 进行解码。对于这些方法,必须限制分子的大小,这严重限制了每个聚合物链的存储容量。
此外,完整的 DNA 链必须按顺序逐个解码,无法直接访问感兴趣的部分。这就像必须读完整本书,而不是只打开相关页面。相比之下,长链 DNA 可以切成随机长度的片段,单独测序,然后通过计算重建原始序列。
首尔国立大学化学系的 Kyoung Taek Kim 和他的团队开发出了一种新方法,可以有效解码分子量大大超过 MS 和 MS2 分析极限的超长合成聚合物链。
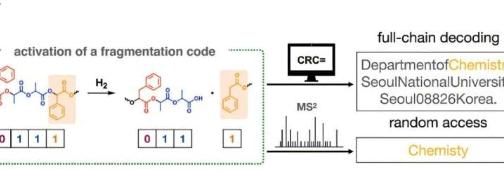
作为演示,该团队将他们的大学地址编码为 ASCII,然后将其与错误检测码(CRC,一种用于确保数据完整性的既定方法)一起转换为二进制代码,即一串 1 和 0。
这个 512 位序列存储在由两种不同单体组成的聚合物链中:乳酸代表 1,苯乳酸代表 0。它们还不规则地包含含有甘醇酸的碎片代码。当化学激活时,这些链会在这些位置断裂。
在他们的演示中,他们获得了 18 个大小各异的片段,可以通过 MS2 测序单独解码。
专门开发的软件最初根据 MS 光谱显示的质量和端基识别碎片。在 MS2 过程中,先前测量的分子离子进一步分解,然后对这些碎片进行分析。
碎片可以根据碎片的质量差异进行测序。借助CRC错误检测码,软件重建了整个链的序列,从而克服了聚合物链的长度限制。
研究团队还能够在不对整个聚合物链(随机访问)进行测序的情况下解码有趣的位,例如其地址代码中的单词“化学”。
通过考虑到其地址的各个部分都按特定顺序排列(部门、机构、城市、邮政编码、国家)并用逗号分隔,他们能够在链中隔离存储所需信息的位置,并仅对相关片段进行排序。
免责声明:本答案或内容为用户上传,不代表本网观点。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。 如遇侵权请及时联系本站删除。
